Введение
При гормоноположительном (HR+) раке молочной железы (РМЖ) часто происходят нарушения в работе сигнального пути PI3K-AKT-mTOR1. Одним из таких нарушений является гиперактивация фосфатидилинозитол-3-киназы (PI3K), которая происходит вследствие появления мутаций в гене PIK3CA. Гиперактивация PI3K связана со злокачественной трансформацией клеток, прогрессированием рака и резистентностью к эндокринной терапии (ЭТ)2–4. Кроме того, опухоли с мутациями в гене PIK3CA продемонстрировали меньшую чувствительность к химиотерапии, а пациенты с такими мутациями — более короткую общую выживаемость (ОВ)5. Активирующие мутации в гене PIK3CA выявляются примерно у 40% пациентов с HR+ HER2- распространенным РМЖ (рРМЖ)6–8.
Алпелисиб является специфическим ингибитором киназы PI3K9 и по результатам исследования III фазы SOLAR-110 рекомендован для лечения пациентов с HR+ HER2- рРМЖ и мутациями в гене PIK3CA, которые ранее получали ЭТ11–13. Рекомендации по лечению РМЖ от Национальной комплексной онкологической сети (NCCN), Американского общества клинической онкологии (ASCO) и Европейского общества медицинской онкологии (ESMO) включают тестирование на наличие мутаций в гене PIK3CA как при выявлении первых метастазов, так и при рецидиве заболевания11–13.
Резюме
- Метод NGS позволяет находить любые изменения в гене PIK3CA, тогда как тестирование методом ПЦР позволяет проверять наличие только конкретных искомых мутаций13.
- Методом NGS дополнительно выявляется 6,7–7,7% пациентов, имеющих мутации в гене PIK3CA, которые не выявляются ПЦР-анализом14,18. Большинство таких мутаций относится к редким подтипам.
- Прямых исследований по эффективности алпелисиба при лечении пациентов с редкими мутациями в гене PIK3CA нет. Анализ данных из реальной клинической практики по всему миру показал, что добавление алпелисиба к фулвестранту может принести пользу пациентам с редкими PIK3CA-мутациями, несмотря на то что таким пациентам алпелисиб назначают реже и в более поздних линиях терапии, чем пациентам с частыми PIK3CA-мутациями18.
Тестирование на мутации: в чем различия методов ПЦР и NGS?
Мутации в гене PIK3CA можно выявлять двумя способами: методом полимеразной цепной реакции (ПЦР) и методом высокопроизводительного секвенирования нового поколения (NGS). Мутации могут возникнуть в любом месте гена PIK3CA. Метод NGS позволяет находить любые изменения в этом гене, тогда как тестирование методом ПЦР позволяет проверять наличие только конкретных искомых мутаций14.
Обычно методом ПЦР проверяют наличие частых мутаций, которые возникают в 11 «горячих точках» гена PIK3CA: H1047L/R/Y (киназный домен, экзон 20), E542K, E545K/A/D/G, Q546E/R (спиральный домен, экзон 9) и C420R (домен C2, экзон 7).
Наличие этих подтипов мутаций, выявленное в образцах опухолевой ткани методом ПЦР, было критерием включения пациентов в когорту с PIK3CA-мутациями в исследовании SOLAR-110. Но когда ретроспективно методом NGS проанализировали 404 образца опухолей пациентов, включенных в это исследование, то удалось дополнительно выявить 31 образец с PIK3CA-мутациями (7,7%), которые изначально не были обнаружены методом ПЦР14. Большинство таких мутаций относилось к редким подтипам.
Частые и редкие мутации в гене PIK3CA
По данным клинического исследования SOLAR-1 и двух ретроспективных исследований реальной клинической практики, самыми распространенными подтипами мутаций гена PIK3CA у пациентов с HR+ HER2- рРМЖ являются H1047R (34,3–37,3%), E545K (17–20,5%) и E542K (11–12,7%)15–17.
В еще одном ретроспективном исследовании мировой клинической практики методом NGS изучили более 30 тыс. опухолевых образцов18. Причем изучали как частые — SOLAR-мутации, которые учитывались в исследовании SOLAR-1, так и другие (редкие) PIK3CA-мутации (рис. 1).
Оказалось, что 34,2% (10869/31765) пациентов имели мутации в гене PIK3CA. Из них у 27,5% (8750/31765) выявлены SOLAR-мутации и у 6,7% (2119/31765) пациентов — другие (редкие) PIK3CA-мутации. При этом одновременно и те, и другие мутации были обнаружены в 3,6% (1146/31765) образцов18.
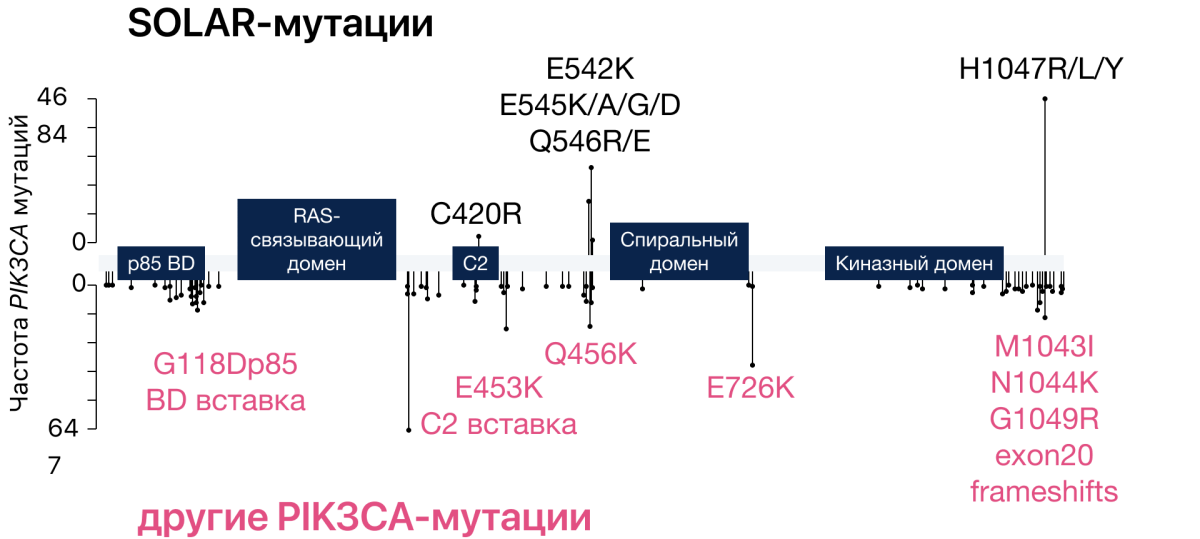
Рисунок 1. Схематическое изображение гена PIK3CA и мутаций в нем: частые SOLAR-мутации (серый цвет, выявляются методами ПЦР и NGS) и редкие PIK3CA-мутации (розовый цвет, выявляются методом NGS)16
Применение алпелисиба при лечении пациентов с частыми и редкими мутациями в гене PIK3CA
Прямых исследований по эффективности алпелисиба при лечении пациентов с редкими мутациями в гене PIK3CA нет. В настоящее время об эффективности алпелисиба у таких пациентов можно судить по результатам ретроспективного анализа данных из реальной клинической практики, в котором было использовано около 1600 историй болезни из анонимной клинико-геномной базы данных Flatiron Health-Foundation Medicine (США)16. Пациенты были разделены на три когорты.
Когорта А:
Пациенты с HR+ HER2- РМЖ с любым типом мутаций в гене PIK3CA (SOLAR-мутации и редкие PIK3CA-мутации). В этой когорте сравнивали эффективность лечения фулвестрантом (n = 124) и комбинацией алпелисиб + фулвестрант (n = 111) в качестве терапии ≥ 2-й линии.
Для когорты A было выявлено увеличение медианы ВБП при добавлении алпелисиба к фулвестранту (6,5 мес. против 4,1 мес., p = 0,027), это продемонстрировало, что в реальной клинической практике алпелисиб дает преимущество пациентам с любым типом PIK3CA-мутаций (а не только носителям SOLAR-мутаций) (рис. 2).
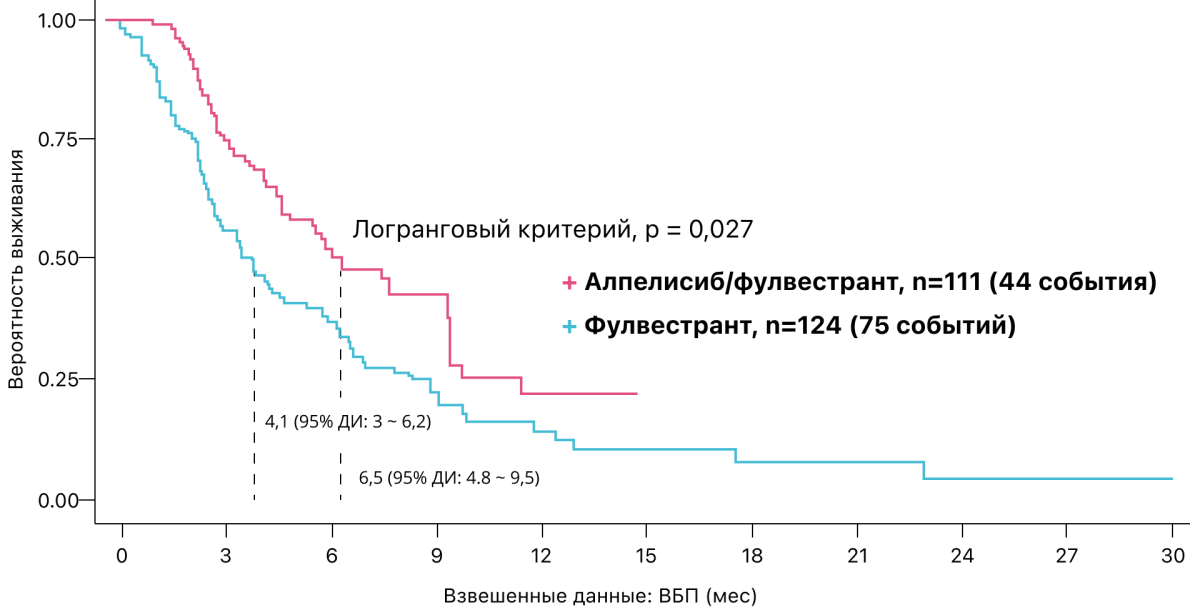
Рисунок 2. ВБП в когорте А пациентов из реальной клинической практики на терапии комбинацией алпелисиб + фулвестрант в сравнении с плацебо + фулвестрант16
Когорта B:
Пациенты с HR+ HER2- РМЖ с любым типом мутаций в гене PIK3CA, получавшие алпелисиб (n = 627). В этой когорте изучали назначение алпелисиба пациентам с SOLAR-мутациями по сравнению с пациентами, у которых выявлены только другие (редкие) PIK3CA-мутации.
- В когорте B алпелисиб получали 39% (202/524) пациентов с SOLAR-мутациями и только 27% (28/103) пациентов с редкими PIK3CA-мутациями.
- Пациенты с SOLAR-мутациями получали лечение алпелисибом раньше, чем пациенты с редкими PIK3CA-мутациями: медиана линий терапии 4L (3L-6L) и 5,5L (4L-8L) соответственно.
Когорта C:
Пациенты с редкими PIK3CA-мутациями, отличными от выявленных в исследовании SOLAR, получавшие алпелисиб (n = 36).
В когорте C у 5 пациентов с редкими мутациями (N345K, Q75E, R38C, G106-108del и N345K/N1044K) зафиксирована ВБП более 6 месяцев, включая пациентов с предшествующей химиотерапией и терапией ингибиторами CDK4/6 (рис. 3).
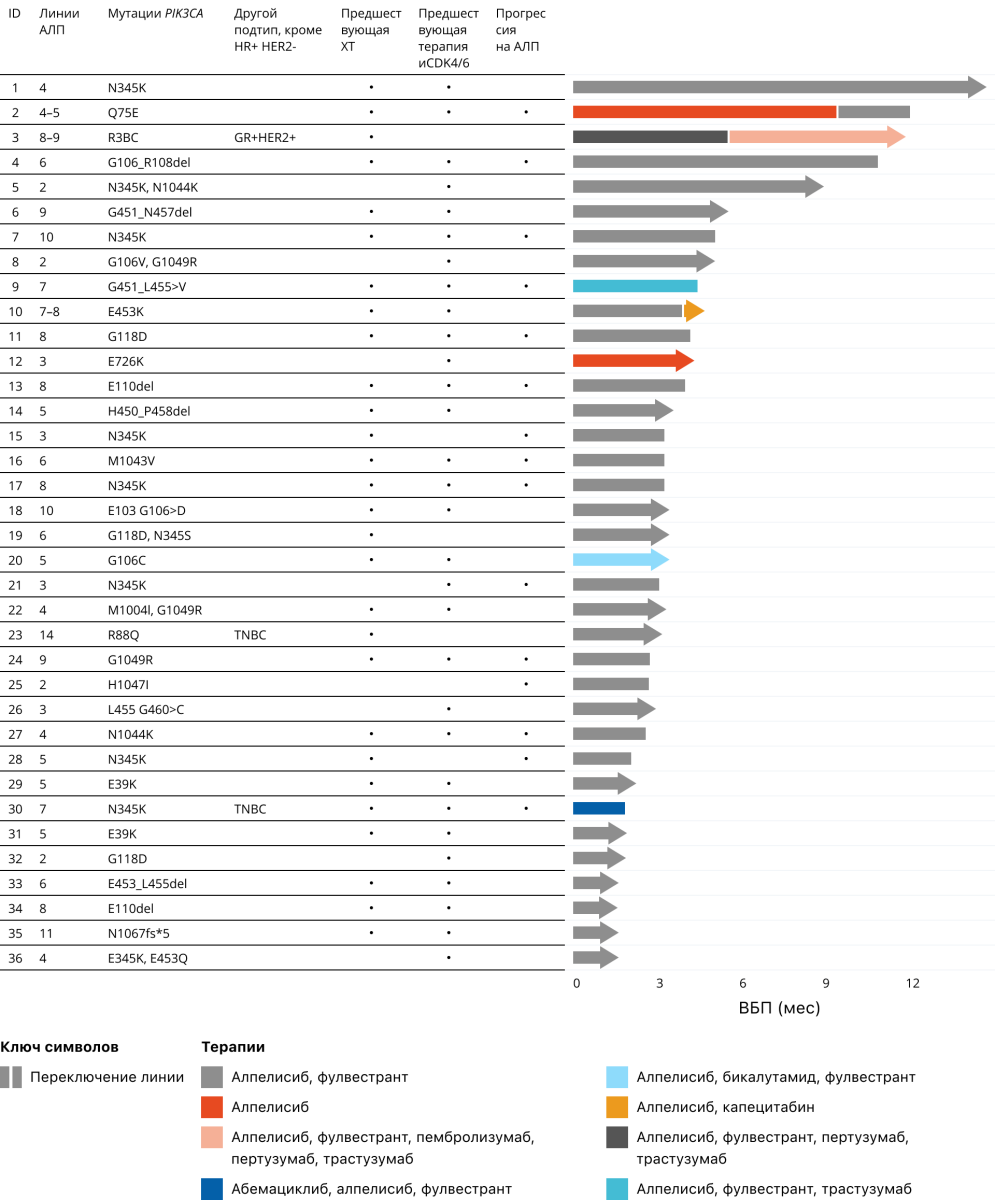
Рисунок 3. Применение алпелисиба в реальной клинической практике при лечении пациентов с редкими PIK3CA-мутациями (когорта С, редкие PIK3CA-мутации)16
Заключение
Мутации в домене C2 (экзон 7), спиральном домене (экзон 9) и киназном домене (экзон 20) являются наиболее частыми мутациями, которые встречаются в гене PIK3CA при HR+ HER2- рРМЖ. Наличие этих мутаций ассоциировано с чувствительностью к алпелисибу, что установлено in vitro и в клиническом исследовании SOLAR-110,17. Данные об использовании алпелисиба при лечении РМЖ с другими (редкими) подтипами PIK3CA-мутаций были получены в ретроспективном исследовательском анализе баз данных.
В связи с отсутствием проспективных рандомизированных исследований по эффективности алпелисиба у пациентов с редкими PIK3CA-мутациями к выбору терапии в реальной клинической практике следует подходить с осторожностью. Однако данные из реальной клинической практики показывают, что добавление алпелисиба к фулвестранту может принести пользу пациентам, у которых есть не только частые, но и редкие PIK3CA-мутации, расположенные за пределами «горячих точек», которые учитывались в исследовании SOLAR-116.
Источники:
- Miller T.W. et al. Phosphatidylinositol 3-kinase and antiestrogen resistance in breast cancer. J Clin Oncol. 2011;29(33): 4452-4461. https://ascopubs.org/doi/10.1200/JCO.2010.34.4879
- Johnston S.R.D. New strategies in estrogen receptor-positive breast cancer. Clin Cancer Res. 2010;16(7): 1979-1981. https://aacrjournals.org/clincancerres/article/16/7/1979/76026/New-Strat...
- Ciruelos Gil E.M. Targeting the PI3K/AKT/mTOR pathway in estrogen receptor-positive breast cancer. Cancer Treat Rev. 2014;40(7): 862-871. https://www.cancertreatmentreviews.com/article/S0305-7372(14)00047-4/fulltext
- Shah P.D., Dickler M.N. Endocrine therapy for advanced breast cancer. Clin Adv Hematol Oncol. 2014;12(4): 214-223. Электронный ресурс: https://www.hematologyandoncology.net/files/2014/04/hoShahDickler4141.pdf (дата последнего доступа 15.06.2022)
- Mosele F. et al. Outcome and molecular landscape of patients with PIK3CA-mutated metastatic breast cancer. Ann Oncol. 2020;31(3): 377-386. https://www.annalsofoncology.org/article/S0923-7534(19)39094-5/fulltext
- Fritsch C. et al. Characterization of the novel and specific PI3k-alpha inhibitor NVP-BYL719 and development of the patient stratification strategy for clinical trials. Mol Cancer Ther. 2014;13(5): 1117-1129. https://aacrjournals.org/mct/article/13/5/1117/91923/Characterization-of...
- Osborne C.K., Schiff R. Mechanisms of endocrine resistance in breast cancer. Annu Rev Med. 2011;62: 233-247. https://www.annualreviews.org/doi/10.1146/annurev-med-070909-182917
- Huang A. et al. Abstract 3749. Single agent activity of PIK3CA inhibitor BYL719 in a broad cancer cell line panel. Cancer Res. 2012;72(8_Supplement): 3749. https://aacrjournals.org/cancerres/article/72/8_Supplement/3749/580934/A...
- Fritsch C. et al. Abstract 3934. Determination of the PI3Kα selective inhibitor alpelisib mechanism of action and efficacy in ER+/PIK3CA mutant breast cancer pre-clinical models. Cancer Res. 2018;78(13_Supplement): 3934. https://aacrjournals.org/cancerres/article/78/13_Supplement/3934/628242/...
- Andre F. et al. Alpelisib for PIK3CA-mutated, hormone receptor-positive advanced breast cancer. N Eng J Med. 2019;380(20): 1929-1940. https://www.nejm.org/doi/10.1056/NEJMoa1813904
- Gennari A. et al. ESMO Clinical Practice Guideline for the diagnosis, staging and treatment of patients with metastatic breast cancer. Ann Oncol. 2021; 32(12): 1475-1495. https://www.annalsofoncology.org/article/S0923-7534(21)04498-7/fulltext
- Burstein H.J. et al. Endocrine treatment and targeted therapy for hormone receptor-positive, human epidermal growth factor receptor 2-negative metastatic breast cancer: ASCO Guideline Update. J Clin Oncol. 2021;39(35): 3959-3977. https://ascopubs.org/doi/10.1200/JCO.21.01392
- Arsenic R. et al. Comparison of targeted next-generation sequencing and Sanger sequencing for the detection of PIK3CA mutations in breast cancer. BMC Clin Phathol. 2015;15:20. https://bmcclinpathol.biomedcentral.com/articles/10.1186/s12907-015-0020-6
- Juric D. et al. Clinical outcomes of alpelisib in hormone receptor-positive, human epidermal growth factor receptor-2-negative advanced breast cancer by next-generation sequencing-detected PIK3CA alteration status and phosphatase and tensin homolog loss: Biomarker analysis from the SOLAR-1 study. Poster Presentation at San Antonio Breast Cancer Symposium — 42nd Annual Meeting;December 10-14, 2019;San Antonio TX. Poster P4-10-04; Cancer Res (2020) 80 (4_Supplement): P4-10-04. https://aacrjournals.org/cancerres/article/80/4_Supplement/P4-10-04/6468...
- Martinez-Saez O. et al. Frequency and spectrum of PIK3CA somatic mutations in breast cancer. Breast Cancer Res. 2020;22(45):1-9. https://breast-cancer-research.biomedcentral.com/articles/10.1186/s13058...
- Razavi P. et al. Real-world natural history data among patients with PIK3CA-mutant and PIK3CA-wild-type advanced breast cancer. In: Proceedings of the 2021 San Antonio Breast Cancer Symposium; 2021 Dec 7-10; San Antonio, TX. Philadelphia (PA): AACR; Cancer Res (2022) 82 (4_Supplement): P1-18-18. https://aacrjournals.org/cancerres/article/82/4_Supplement/P1-18-18/6804...
- Rajadurai P. et al. PIK3CA Registry: A noninterventional, descriptive, retrospective cohort study of PIK3CA mutations in patients with hormone receptor-positive (HR+), human epidermal growth factor receptor 2-negative (HER2-) advanced breast cancer (ABC). In: Proceedings of the 2021 San Antonio Breast Cancer Symposium; 2021 Dec 7-10; San Antonio, TX. Philadelphia (PA): AACR; Cancer Res (2022) 82 (4_Supplement): P5-13-25. https://aacrjournals.org/cancerres/article/82/4_Supplement/P5-13-25/6813...
- Rugo H.S. et al. Real world (rw) clinical outcomes on alpelisib (ALP) in patients (pts) with breast cancer (BC) and PIK3CA mutations (PIK3CAm). J Clin Oncol. 2021;39(15_suppl): 1068-1068. https://ascopubs.org/doi/10.1200/JCO.2021.39.15_suppl.1068
11249630/ALP/web/08.24/0

